장내 미생물 등 메타유전체(특정 환경의 모든 미생물 유전체 총합) 빅데이터를 쉽고 빠르게 분석할 수 있는 도구가 개발됐다. 마이크로바이옴 연구 문턱이 낮아질 전망이다.
한국연구재단(이사장 홍원화)은 김지현 연세대 교수팀이 권순경 경상국립대 교수 등과 대용량 메타유전체 정보 기능 분석을 표준화·최적화한 통합 파이프라인 '메타펀(metaFun)'을 개발했다고 26일 밝혔다.
인체 마이크로바이옴은 우리 몸 속 미생물의 집합이다. 그 군집의 구조·기능을 들여다볼 수 있는 메타유전체 분석 기술은 마이크로바이옴 연구 핵심으로 자리 잡았다. 하지만 분석 과정이 매우 복잡하고, 사용 프로그램이나 설정값에 따라 결과가 크게 달라지는 문제가 있다. 프로그램 사용 및 코딩에 익숙하지 않은 임상·생물학 연구자들이 특히 어려움을 겪었다.
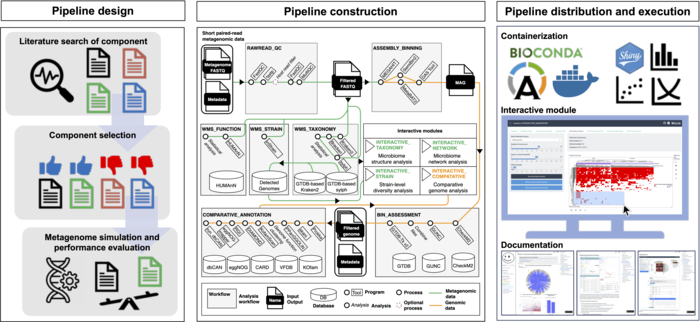
이에 연구진은 메타유전체 분석 전 과정에 대해 표준화하고 최적화한 데이터 해석 환경을 제공하는 분석 도구를 설계했다. 인체 장내, 식물 뿌리 토양, 해양 등 다양한 환경의 메타유전체 데이터를 활용한 대규모 테스트를 통해 최적의 분석 조건을 도출했다.
또 서로 다른 컴퓨터 분석 환경에서도 같은 결과를 얻을 수 있도록 최신 워크플로우 관리 기술(데이터 분석의 여러 단계가 끊김없이 수행되도록 관리하는 기술)과 컨테이너 기술(소프트웨어 실행 요소들을 패키지화한 가상화 기술)을 적용했다.

이렇게 개발한 메타펀은 염기서열 데이터 품질 검사부터 미생물 종류 분석, 기능 분석, 균주 수준의 세밀한 분석, 유전체 조립과 분류, 유전체 품질 평가, 비교유전체학 분석에 이르는 총 7개 분석 단계를 효율적으로 수행한다.
또 분석 결과를 활용해 실시간 데이터 해석과 시각화를 지원하는 4개 대화형 모듈을 오픈소스로 제공해 비전문가도 코딩 기술 없이 결과를 이해할 수 있도록 했다.
관련 논문 제1저자인 이현권 박사는 “대장암 환자와 건강인의 장내 미생물 데이터를 이용한 메타펀 성능 검증에서 환자군과 건강인 간 뚜렷한 차이를 확인해 실제 연구에 활용 가능한 도구임을 입증했다”고 밝혔다.
김지현 교수는 “개별 미생물 종류의 변화부터 군집 간 상호작용 네트워크, 나아가 균주 수준의 미세다양성까지 다층적으로 분석할 수 있어, 마이크로바이옴과 질병의 연관성, 환경에서의 역할 등을 보다 입체적으로 규명할 수 있을 것”이라고 설명했다.
과학기술정보통신부와 한국연구재단이 추진하는 바이오·의료기술개발사업 및 중견연구사업 등의 지원을 받은 이번 연구 성과는 '장내 미생물(Gut Microbes)'에 지난 13일 온라인 게재됐다.
김영준 기자 kyj85@etnews.com












































