한국과학기술정보연구원(KISTI)이 슈퍼컴퓨터를 활용한 독감 바이러스의 변이 예측에 도전장을 내밀었다.
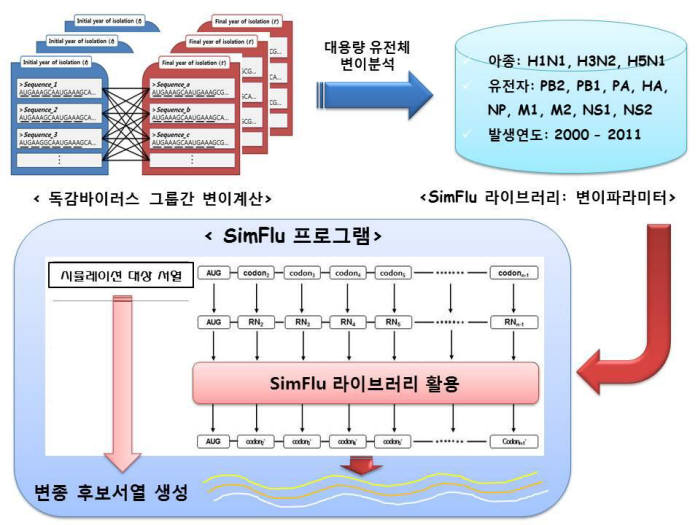
한국과학기술정보연구원(원장 박영서) 국가슈퍼컴퓨팅연구소 고성능바이오컴퓨팅팀은 변이 예측을 위해 심플루(SimFlu)를 개발했다. 심플루는 변이 예측 시뮬레이션 프로그램이다.
독감 바이러스는 빠른 속도로 다양한 종류의 변이체를 생산하기 때문에 변종에 대한 예측이 어려운 것으로 알려져 있다. 매년 많은 사람들이 독감 백신을 접종하고 있지만 변종이 발생할 경우 속수무책으로 당하기 쉽다.
이에 대응하기 위해 KISTI는 서울대학교 보건대학원과 3년간 공동연구를 통해 전 세계 5만6000여개의 H1N1, H5N1 및 H3N2 아종에 대한 유전체 서열을 수집해 변이되는 패턴을 계산하는 데 성공했다. 이 패턴을 활용하면 컴퓨팅 환경에서 미리 발생 가능한 변종 바이러스 후보군을 생성해 볼 수 있게 된다.
실제로 지난 1999년 발생한 독감 바이러스 서열을 대상으로 2008년 시뮬레이션을 수행한 결과, 그 다음해인 2009년 발병한 신종 플루와 유사성이 있는 후보 서열들을 얻는 데 성공했다.
연구진은 심플루 프로그램 PC버전은 웹을 통해 서비스 중이다. 변이 계산 모듈에 가시화 기능을 추가한 버전은 이르면 12월께 공개할 계획이다.
안인성 KISTI 고성능바이오컴퓨팅팀장은 “KISTI의 슈퍼컴퓨팅 인프라와 시뮬레이션 원천기술을 바탕으로 갑작스러운 신종 감염병 창궐을 예측·대응할 수 있는 초석을 마련했다”고 밝혔다.
대전=박희범기자 hbpark@etnews.com